| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- XGBoost
- Machine Learning
- Explainable AI
- lime
- Gradient Tree Boosting
- data science
- deep learning
- Back-propagation
- Gradient Boosting Machine
- Today
- Total
Kicarussays
[논문리뷰/설명] Forecasting adverse surgical events using self-supervised transfer learning for physiological signals 본문
[논문리뷰/설명] Forecasting adverse surgical events using self-supervised transfer learning for physiological signals
Kicarus 2022. 3. 8. 23:58새로운 연구디자인을 찾으려 npj Digital Medicine에서 논문들을 살펴보던 중, SHAP을 개발하신 이수인 교수님의 논문을 발견했습니다.
본 논문은 PHASE (PHysiologicAl Signal Embeddings) 라는 방법을 새롭게 제시하여 EHR 데이터와 생체신호 데이터를 활용하여 hypoxemia(저산소혈증), hypocapnia(저탄산혈증), 저혈압, 고혈압, phenylephrine (저혈압 조절에 사용하는 약물) 그리고 epinephrine (마취를 돕는 약물) 까지 6가지 독립적인 수술 부작용을 예측한 연구를 소개합니다.
시작하겠습니다!
논문링크: https://www.nature.com/articles/s41746-021-00536-y
Forecasting adverse surgical events using self-supervised transfer learning for physiological signals - npj Digital Medicine
Five perioperative outcomes from three hospital datasets We are interested in forecasting important outcomes associated with surgical morbidity. The first is hypoxemia (i.e., low blood oxygen level), a historically important risk factor associated with ane
www.nature.com
Introduction
- 수술 부작용은 적절한 예측 모델을 활용하여 방지할 수 있음
- 예측 모델의 성능은 training data의 활용 가능성에 달려 있는데, 환자 개인정보 이슈로 인해 병원 데이터들은 반출될 수 없음
- 충분하지 못한 데이터로 학습을 하는 문제를 다루기 위해 전이학습(Transfer Learning)을 활용할 수 있음
- 전이학습의 한 방법으로, 이미지나 시계열 데이터로부터 정형 feature를 추출하여 학습하는 deep embedding model 이 있음
- 본 논문의 PHASE (PHysiologicAl Signal Embeddings) 는 deep embedding model 을 학습하여 수술 부작용을 예측함
- PHASE는 기존 방법보다 예측 정확도를 향상시켰고, 전이학습에 활용할 수 있음
- self-supervised LSTM을 활용하여 생체신호 데이터의 정보를 포함하고 있는 embedding vector를 가져오고, 이를 환자의 나이, 키 등의 정보를 포함하고 있는 EHR 데이터와 결합(concatenate)한 후, GBT (Gradient Boosting Tree) 모델로 학습하여 수술 후 부작용을 예측함
- 충분한 데이터가 있는 병원에서 PHASE 모델을 학습하고, 이를 전이학습을 활용하여 다른 병원에 맞게 재학습시킬 수 있음
- 본 연구에서는 AIMS에서 2개 병원의 수술실 데이터와 MIMIC-III의 중환자실 데이터를 활용하여 학습하였고, 이를 다른 병원들에서 전이학습을 통해 활용하게끔 한다는 것이 PHASE의 취지임

위 그림은 본 논문의 연구를 요약한 그림입니다. a는 생체신호 데이터와 EHR 데이터를 활용하여 부작용을 예측하는 과정을 보여줍니다. b는 PHASE를 통해 이미 만들어진 모델을 target 병원에서 전이학습을 통해 활용하는 과정을 보여줍니다. c는 PHASE에서 생체신호 데이터를 self-supervised learning을 통해 임베딩하는 방식을 보여주고 있습니다.
Methods
Dataset
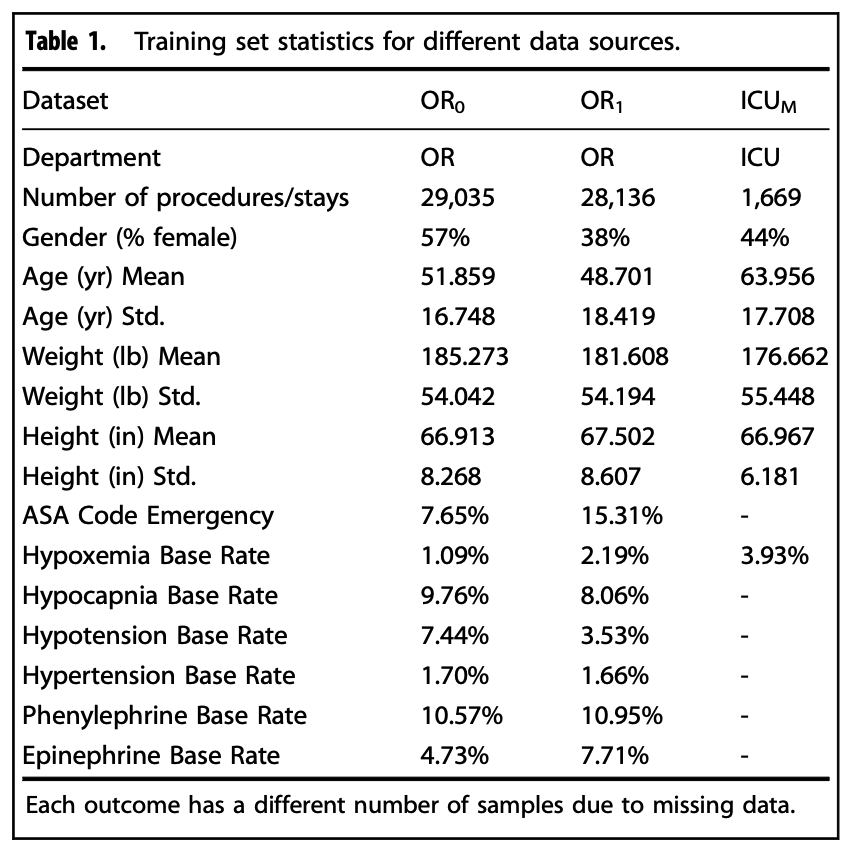
OR은 수술실, ICU는 중환자실을 의미합니다. 본 연구에서는 6개 항목의 EHR 데이터(static)와, 15개의 생체신호(dynamic)를 input으로 사용했다고 언급하고 있습니다. 또한 평가 metric으로는 각 예측 대상에 대해 AUROC를 사용했습니다.
Variables
6개 항목의 static, 15개 항목의 dynamic 변수들을 사용하고 있습니다. 이 데이터를 본 연구에서는 아래와 같이 표현합니다.
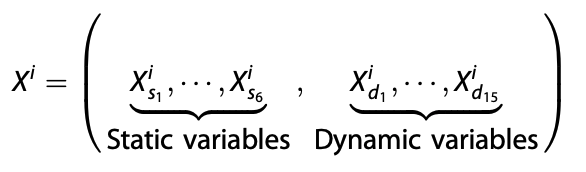
Static:
- Height,
- Weight,
- ASA Code,
- ASA Code Emergency,
- Gender,
- Age
Dynamic:

Outcomes:
최종적으로 예측하고자 하는 것은 6개의 수술 후 부작용이고, 이는 binary 변수로 설정합니다.

Dynamic Embedding
PHASE는 dynamic 변수들에 해당하는 생체신호 데이터를 임베딩하고, 임베딩 벡터를 static 변수들과 결합한 데이터를 GBT(Gradient Boosting Tree) 모델에 입력하여 outcome을 예측합니다.
dynamic 변수의 경우 동일한 형태를 가져오기 위해 예측 시점 이전 60분간의 신호 를 활용합니다.
임베딩 벡터를 산출하는 방법으로는 다음과 같습니다.

- raw: 데이터 그대로 활용
- ema: 이동평균 활용
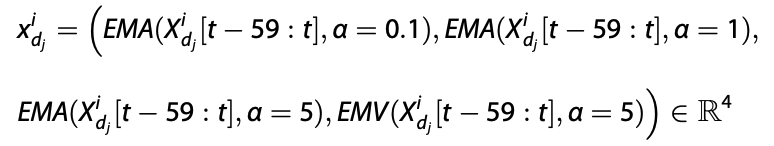
- rand: random initialize LSTM의 마지막 Hidden layer 활용
- auto: autoencoder LSTM의 마지막 Hidden layer 활용
- next: 이후 신호 데이터를 예측하는 LSTM의 마지막 Hidden layer 활용
- min: 이후 신호 데이터의 최소값을 예측하는 LSTM의 마지막 Hidden layer 활용
- hypo: target outcome을 예측하는 LSTM의 마지막 Hidden layer 활용
본 연구에서는 LSTM의 hidden layer의 size를 200으로 설정하여 LSTM을 활용한 임베딩 벡터의 크기는 200입니다.
Fine-tuned Embedding
본 연구는 2개 수술실(OR)의 데이터를 활용하고 있습니다. dynamic embedding 과정에서 fine-tuning을 위해 한 수술실 데이터로 학습시킨 LSTM embedding 모델을 초기 모델로 설정하고 다른 수술실의 데이터로 학습시켰습니다.
Results
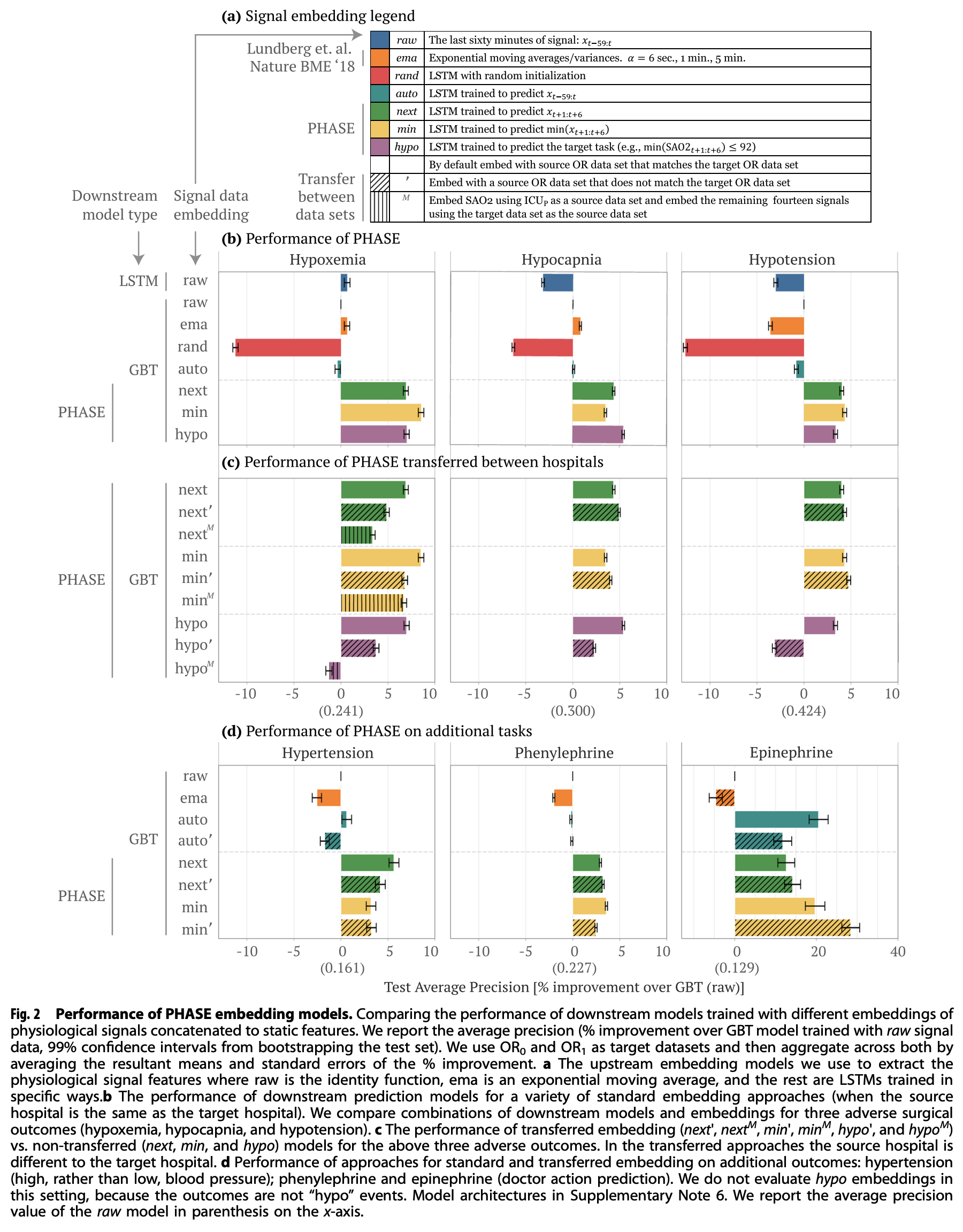
본 연구에서 Downstream model은 GBT에 해당하고, Upstream model은 dynamic embedding을 수행하는 LSTM에 해당합니다. 다음 결과는 dynamic embedding 방식에 따른 결과를 비교한 것입니다.
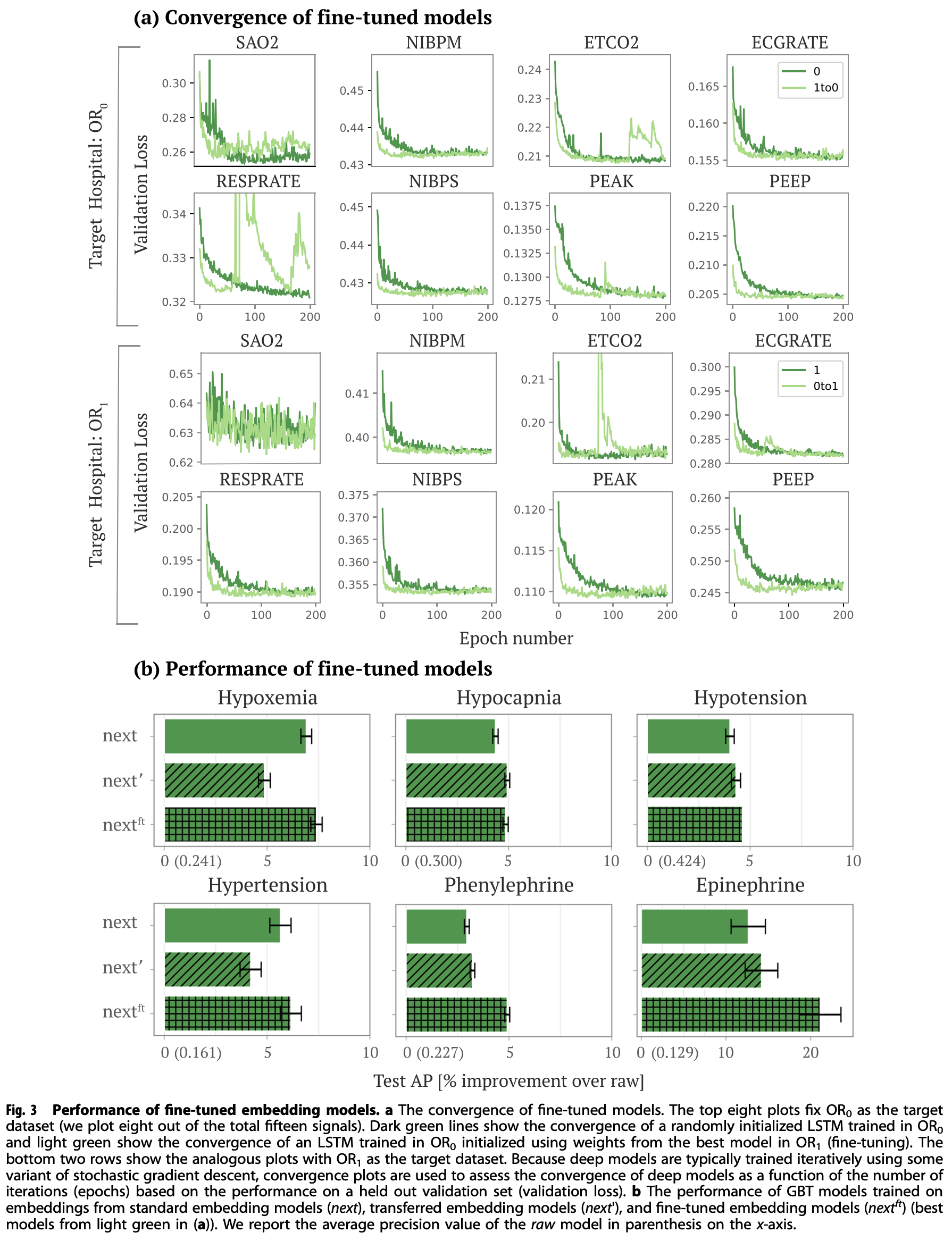
Fine-tuning에 따라 개선되는 결과를 보여주고 있습니다.
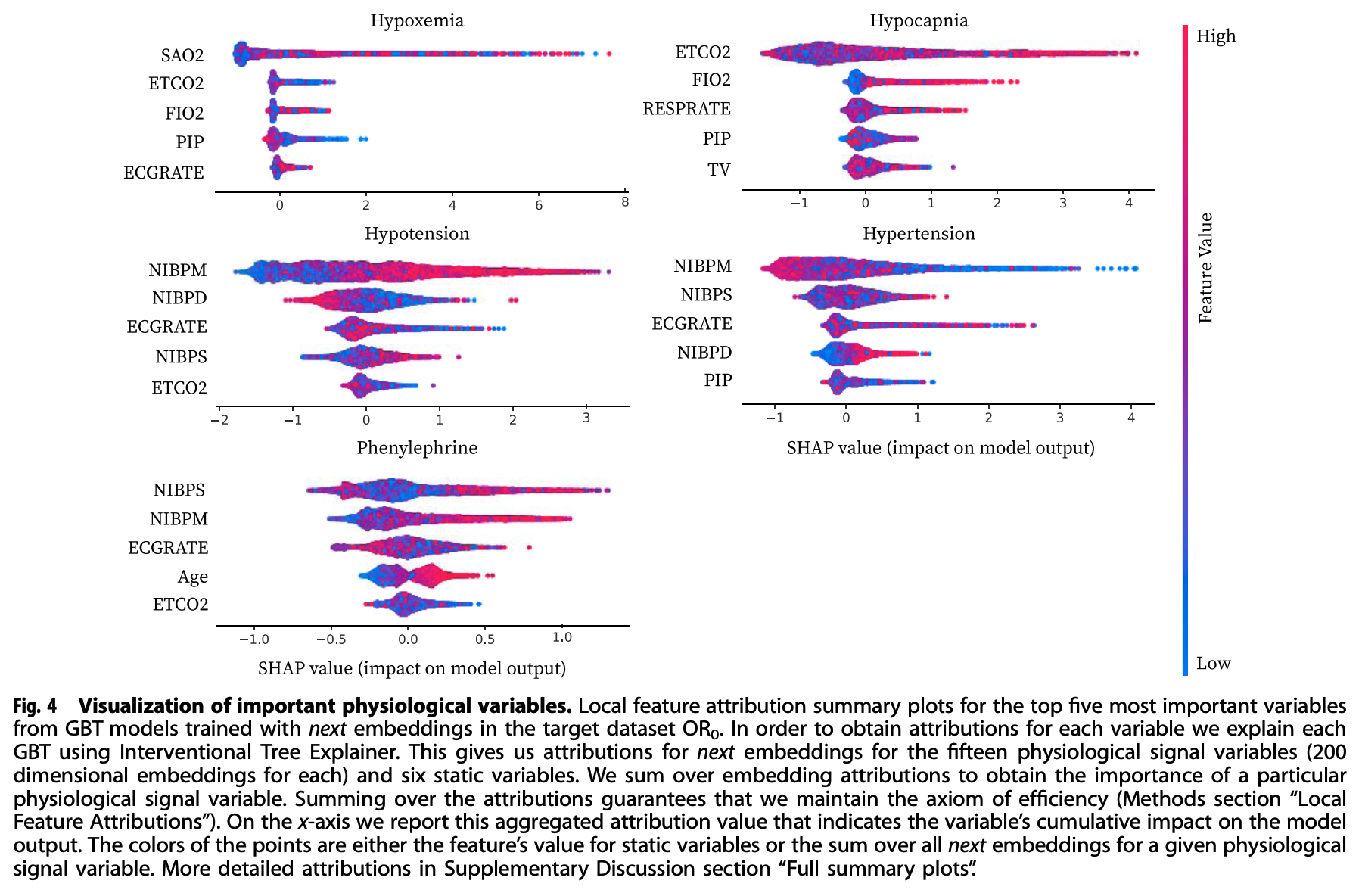
최종 예측모델에 SHAP을 적용하여 변수 중요도를 산출하였습니다.
Conclusion
정형(static) 데이터와 생체신호(dynamic) 데이터를 적절히 결합하여 좋은 결과를 이끌어낸 연구였습니다. SHAP을 활용하여 주요 변수를 찾아내어 설명력을 더했다는 점에서 높은 수준의 연구인 것 같습니다. 또한 큰 규모의 데이터를 가지고 있는 기관에서 학습시킨 모델과 전이학습을 활용하여 범용적으로 활용할 수 있는 가능성을 제시한 부분이 의미가 있었습니다.
감사합니다!

